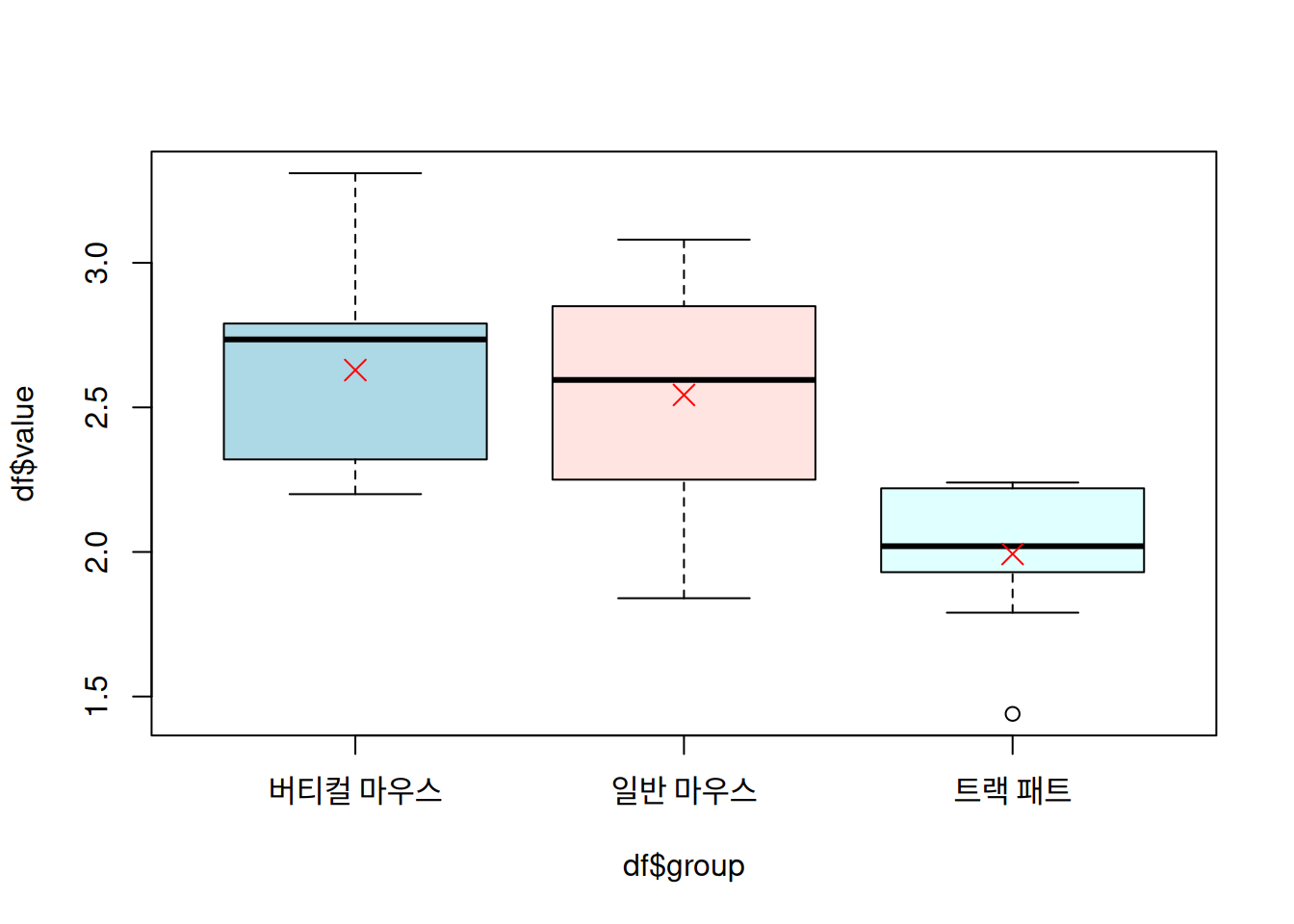
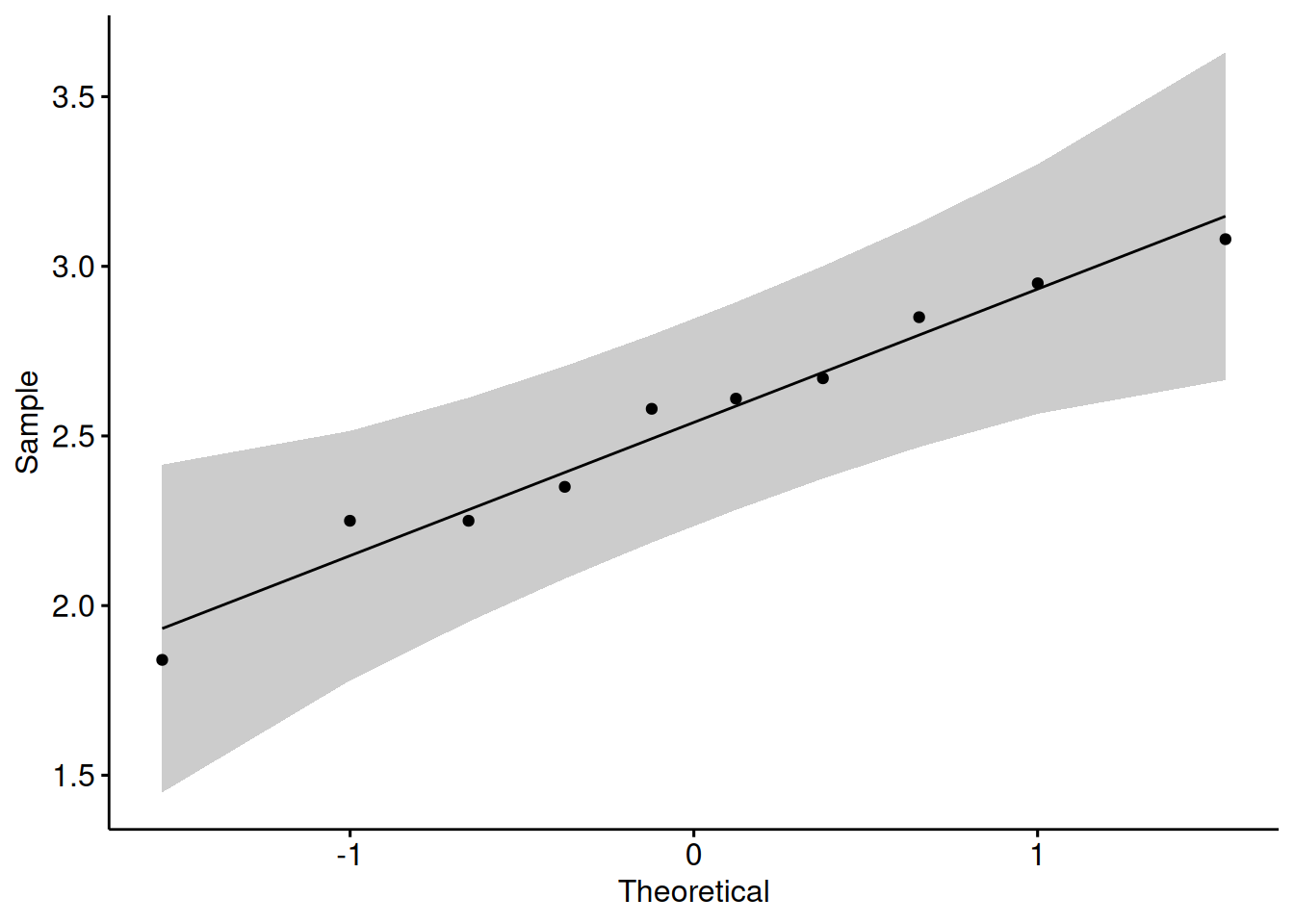
val1 <- c(1.84, 2.67, 2.61, 2.95, 2.25, 2.35, 2.85, 2.58, 3.08, 2.25)
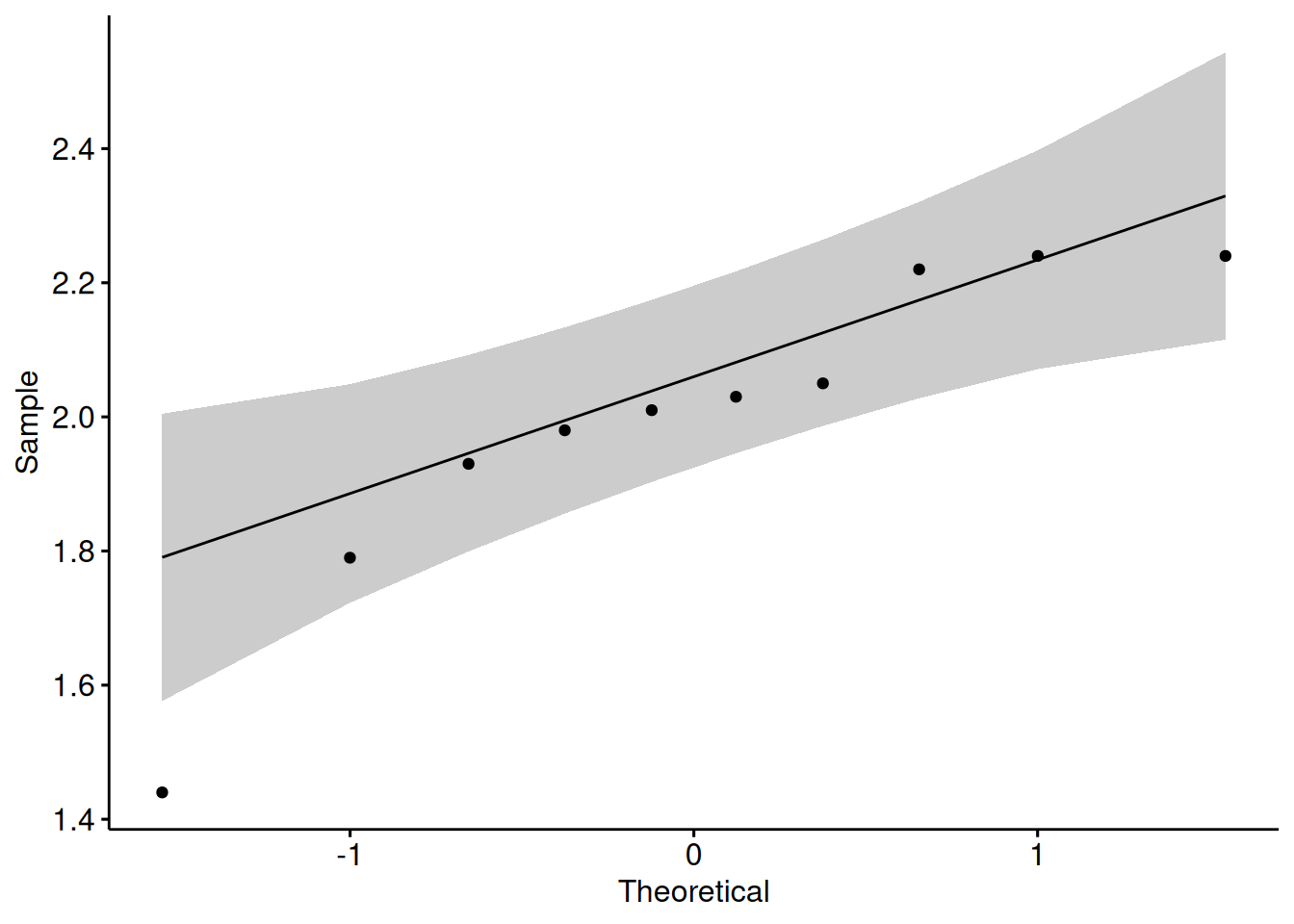
val2 <- c(1.79, 1.98, 2.01, 1.93, 2.24, 2.03, 2.22, 1.44, 2.24, 2.05)
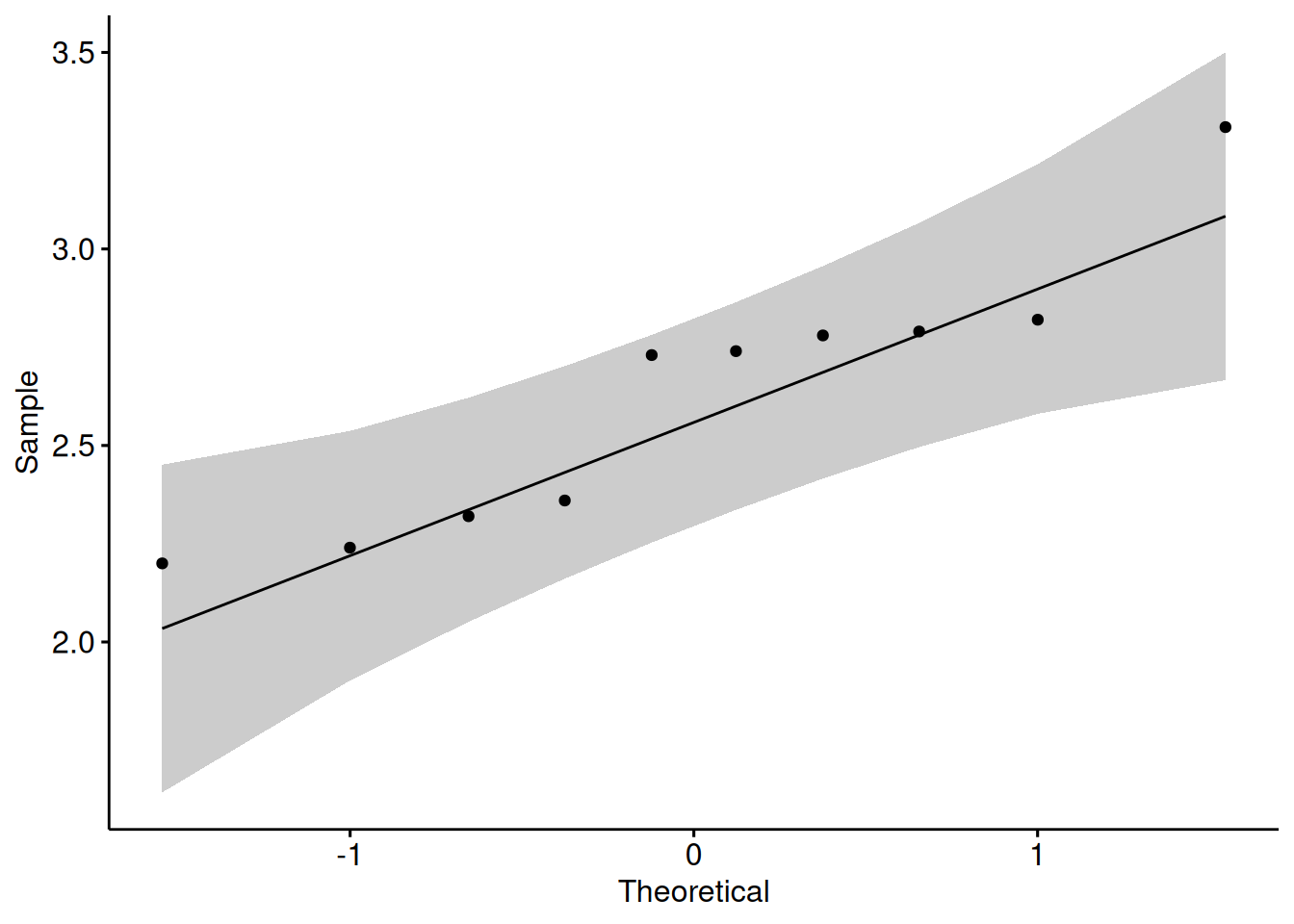
val3 <- c(3.31, 2.24, 2.74, 2.36, 2.79, 2.20, 2.32, 2.82, 2.73, 2.78)
df <- data.frame(
group = c(rep("일반 마우스", 10), rep("트랙 패트", 10), rep("버티컬 마우스", 10)),
finger = c('왼중지', '왼검지', '오른약지', '왼약지', '왼엄지', '오른약지', '오른새끼', '오른엄지', '왼중지', '왼새끼', '오른중지', '오른엄지', '오른중지', '오른검지', '왼엄지', '왼검지', '오른새끼', '오른검지', '왼엄지', '오른중지', '왼검지', '왼새끼', '오른검지', '왼약지', '오른새끼', '오른엄지', '왼약지', '왼중지', '왼새끼', '오른약지'),
value = c(val1, val2, val3)
)
df group finger value
1 일반 마우스 왼중지 1.84
2 일반 마우스 왼검지 2.67
3 일반 마우스 오른약지 2.61
4 일반 마우스 왼약지 2.95
5 일반 마우스 왼엄지 2.25
6 일반 마우스 오른약지 2.35
7 일반 마우스 오른새끼 2.85
8 일반 마우스 오른엄지 2.58
9 일반 마우스 왼중지 3.08
10 일반 마우스 왼새끼 2.25
11 트랙 패트 오른중지 1.79
12 트랙 패트 오른엄지 1.98
13 트랙 패트 오른중지 2.01
14 트랙 패트 오른검지 1.93
15 트랙 패트 왼엄지 2.24
16 트랙 패트 왼검지 2.03
17 트랙 패트 오른새끼 2.22
18 트랙 패트 오른검지 1.44
19 트랙 패트 왼엄지 2.24
20 트랙 패트 오른중지 2.05
21 버티컬 마우스 왼검지 3.31
22 버티컬 마우스 왼새끼 2.24
23 버티컬 마우스 오른검지 2.74
24 버티컬 마우스 왼약지 2.36
25 버티컬 마우스 오른새끼 2.79
26 버티컬 마우스 오른엄지 2.20
27 버티컬 마우스 왼약지 2.32
28 버티컬 마우스 왼중지 2.82
29 버티컬 마우스 왼새끼 2.73
30 버티컬 마우스 오른약지 2.78